01
良渚实验室沈宁课题组围绕“组学与精准医学分析算法开发与应用”开展临床转化密切相关的研究,运用生物信息学数据整合分析与人工智能算法,并结合实验筛选平台进行药物研发与精准治疗。
课题组目前有多项具有重要应用价值的课题正在推进,与著名医学专家主导的实验室有合作关系,诚招具有实验生物、计算生物背景的博士后和研究助理。
详细招聘信息见:https://person.zju.edu.cn/shenning
简历投递(有意者请将个人简历等材料发送至):shenningzju@zju.edu.cn
02
沈宁博士,浙江大学良渚实验室百人计划研究员,博士生导师。沈宁课题组围绕“组学与精准医学分析算法开发与应用”开展与临床转化密切相关的研究,运用生物信息学数据整合分析与人工智能算法,并结合实验筛选平台进行药物研发与精准治疗。在公司工作期间,作为核心成员参与了面肩肱型肌营养不良症(Facioscapulohumeral muscular dystrophy, FSHD)的药物研发,负责了该药物的作用机制研究与临床二期试验生物标志物筛选;作为共同发明人申请了20余项国际专利,已获得美国、加拿大、日本等国家的7项专利授权。目前,该药物已获得美国FDA新药申请(Investigational New Drug, IND)、孤儿药资格认定(Orphan Drug Designation)和快速审批通道许可(Fast Track Designation),已在临床三期试验开展阶段。同时,沈宁研究员在Genome Medicine、PNAS、Cell Systems等杂志作为一作/通讯发表多篇论文。
课题组主要从事组学药理学、生物信息学研究。课题组围绕“疾病基因组到转录组等表型组学过程中的模式识别问题”开展多维度研究,聚焦疑难未诊断疾病,运用生物信息学数据整合分析与人工智能算法,并结合实验筛选平台进行药物研发与精准治疗。主要研究方向如下:1、通过整合大规模基因组转录组等多组学数据进行计算生物分析与算法开发,研究不同疾病中基因组表达调控的生物机制,针对癌症及其他疾病进行药物靶点预测、筛选,以及进行精准医疗相关的生物标志物分析与预测。2、针对罕见遗传病等疾病开展AI引导下设计的生物大分子、小分子治疗方法的药物研发。3、开发基于计算生物学的疾病诊断方法以及对未诊断遗传病患者进行基因诊断分析。
03
招聘岗位—博士后
1. 岗位要求
1)实验生物
近期(或即将)获得生物医学相关领域的博士学位。
有扎实的分子生物学、细胞生物学研究经验,熟悉细胞培养或微生物培养;熟悉PCR、Western、免疫沉淀和NGS等分子生物学技术。
曾以第一作者发表SCI论文,有一定的独立工作能力。
有责任心与团队合作精神,拥有较为良好的英语沟通和写作能力。
熟悉多组学测序实验(WGS, scRNA-seq, ChIP-seq, ATAC-seq等)或有药物研发经验的优先考虑。
2)计算机生物学、生物信息学
近期(或即将)取得生物信息、生物医学、计算机、数学、物理或统计相关领域的博士学位(先后顺序无优先级排序)。
具有扎实的编程能力以及概率统计、机器学习方面知识。
曾以第一作者发表SCI论文,有一定的独立工作能力。
对于计算生物学有研究兴趣,有责任心与团队合作精神,拥有较为良好的英语沟通和写作能力。
具有生物信息学和基因测序大数据分析及算法开发相关研究背景者优先考虑。
2. 薪酬待遇
参照浙江大学良渚实验室博士后管理规定,提供专业的科研指导和具有竞争力的待遇(年薪26万起),提供五险一金,其他福利待遇根据应聘人员资历从优提供。
支持申报博士后科学基金以及其他国家与地方科研项目。课题组与美国、加拿大、欧洲多个国际知名高校课题组有合作联系,表现优异者有机会申请国际交流或联合培养。
博士后出站优秀者有机会申报特聘研究员或副研究员,入职杭州工作者可获得政府资助的安家费及各种人才补贴。
招聘岗位—科研助理
1. 岗位要求
1)计算生物学、生物信息学
近期获得生物信息、生物医学、计算机、数学、物理或统计等相关领域的本科及以上学位。
具有熟练的编程能力。
工作细心,踏实肯干,有研究兴趣、责任心和上进心。
2)实验生物
近期获得生物医学相关领域的硕士及以上学位,具有分子生物学基因组学等相关的研究背景。
具有扎实的分子实验与细胞实验基础,熟悉PCR、Western、免疫沉淀和NGS等分子生物学技术。
工作细心,踏实肯干,有研究兴趣、责任心和上进心
2. 薪酬待遇
根据浙江大学良渚实验室相关规定以及申请人工作能力,实验室将提供在国内外具有竞争力的薪酬待遇以及科研条件,享受五险一金等相关福利(具体待遇面议)。
课题组提供积极的工作环境与灵活的工作制度,为成员发展提供最大可能的帮助。
04
请申请人提供详细的个人简历,包括学习工作经历、主要研究工作内容、代表性论文论著清单、获奖情况等,将材料电子版发送至电子邮箱:shenningzju@zju.edu.cn,邮件标题请注明“姓名_申请博士后”或者“姓名_申请科研助理”。博士后申请人需提供2-3个推荐人信息。初审通过者将安排面试。长期有效,直至招聘满相应岗位为止,应聘材料将予以保密。
05

开篇图注:沈宁课题组学生作图,展现练轻功的少年准确找到还未盛开的融合荷花,暗喻Anchored-fusion技术高灵敏检测到融合基因。
浙江大学良渚实验室沈宁团队Cell Reports Methods 发布基于RNA序列检测融合基因的方法
基因融合是癌症产生的最重要的驱动因素。基因融合的及时诊断对癌症患者的肿瘤分型、分级、分期,有针对性的治疗方法的设计以及疾病进展及预后的评估有着至关重要的意义。基于转录组测序数据(RNA-seq)进行识别是融合基因诊断的重要方法,相对于传统方法,它具有成本低、效率高的特点。然而,基于RNA-seq的方法的灵敏度远低于传统方法,难以应用在测序深度较低或者融合基因转录水平较低的情况,因此其尚不能在临床融合基因检测中作为主流方法使用。
现有基于RNA-seq方法的灵敏度主要受到以下两条限制,一是融合基因支持证据的遗漏。基因组中广泛存在同源序列和短同源序列。当应匹配到目标融合基因的短读长序列(reads)被匹配到同源序列上时,该短读长序列就不能作为目标融合基因的支持证据,使检出的支持证据减少。因此,匹配到基因组上多处位点的短读长序列会被过滤掉而不能作为支持证据。二是测序过程中会产生虚假融合基因,为了减少检出的假阳性融合基因数量,现有工具设置了支持证据数目阈值,若融合基因的支持证据数目未达到阈值,将被视为假阳性而被过滤。这种设定阈值的方法同时也会过滤掉一些支持证据较少的真正融合基因。

2024年3月25日,良渚实验室沈宁研究员、浙江大学计算机学院王海帅研究员,硕士生原溪璐等在Cell Reports Methods上发表了题为Anchored-fusion enables targeted fusion search in bulk and single-cell RNA sequencing data的研究论文,开发了Anchored-fusion算法,以解决现有基于RNA-seq的融合基因检测方法灵敏度不足的问题。
Anchored-fusion算法由两个重要部分组成。一是将输入测序序列中所有的短读比对到用户给出的目标基因转录序列,由此可以保留所有的短读长序列,包括那些可能会被比对到其同源序列的部分。这一方法以最大可能寻找融合基因的支持证据,因此有助于提升融合基因检测的灵敏性。二是Anchored-fusion使用具有局部-全局层级结构的深度神经网络,并使用真实融合基因和虚假人工嵌合序列作为正负样本进行训练,因此能够得到更准确的分类结果。同时,这种方法能够避免依靠设置支持证据数目阈值而过滤掉那些支持证据较少的真实融合基因,提高工具发现目标融合基因的灵敏性。
Anchored-fusion是针对用户指定的锚定基因X,寻找基因X参与的融合基因,其输入数据类型为RNA双端测序短读序列。Anchored-fusion首先将待测RNA-seq数据集中的全部短读比对到用户提供的靶向基因X的参考序列上,能够最大程度地保留可能由基因X转录产生的RNA-seq,步骤避免了X的融合基因产生的RNA-seq被比对到X的同源序列上而被过滤掉,对提升方法的灵敏度十分有效。其次,Anchored-fusion选择只有一条序列比对到锚定基因X上的短读对,并将不能比对到基因X上的另一条序列比对到全基因组上。这部分序列比对到的基因组成候选融合伴侣基因集Y。接下来,Anchored-fusion将失配的短读序列进行局部比对,在融合伴侣基因集Y中找出具有明确融合位点的基因组成候选融合基因。由一对同源基因组成的候选融合基因或跨越-分割读取片段数目极不平衡的融合基因将被过滤。
Anchored-fusion使用深度神经网络区分真实融合和虚假融合。研究人员设计了一个称为层级结构的蒸馏学习模型(Hierarchical View Learning and Distillation,HVLD)。HVLD的输入包含融合位点的融合基因局部序列,经由one-hot编码后输入由CNN组成的两个局部信息提取模块和一个由Transformer encoder组成的全局信息提取模块,分别提取融合基因序列的局部信息,和所有局部信息间的关系。最终HVLD模型将输出该融合基因是真实融合的概率。
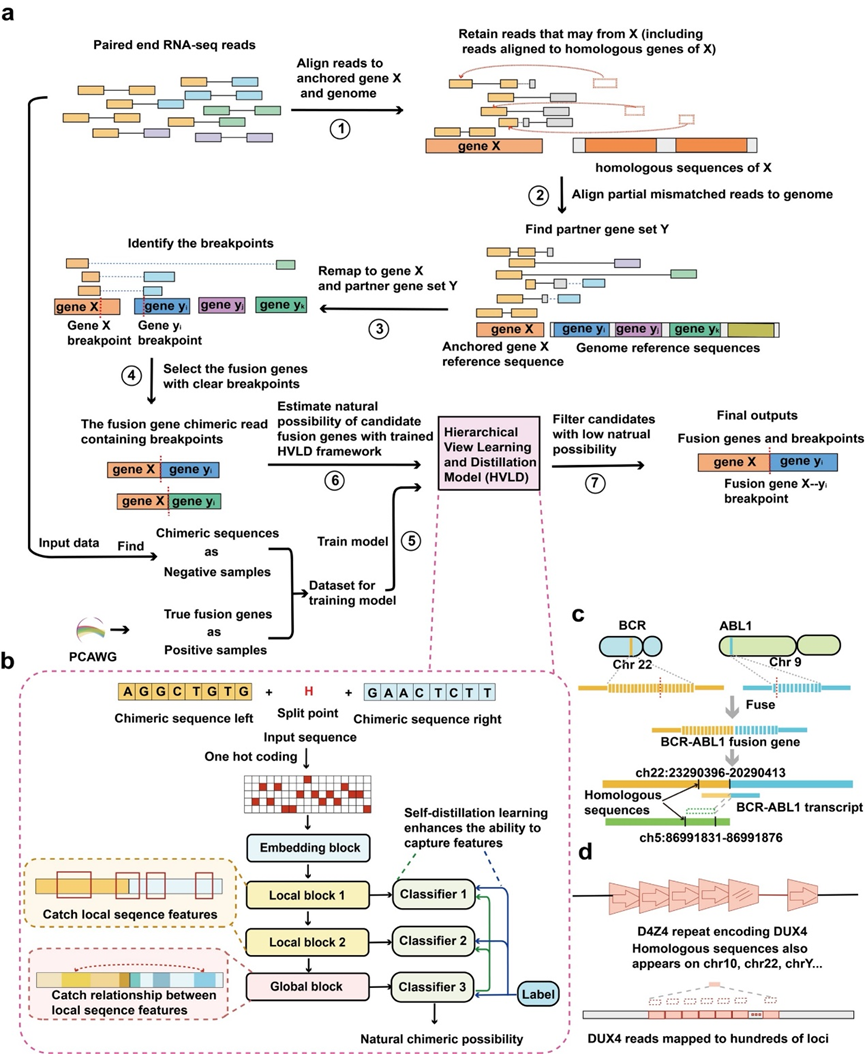
Anchored-fusion及HVDL模型的流程图
研究人员首先选择了六个含有融合基因的肿瘤细胞系及其相应的融合基因,检查了Anchored-fusion在分辨真实融合和虚假融合的能力,以及在模拟和真实的细胞系bRNA-seq 数据中检测融合基因的能力。
研究人员从该六个肿瘤细胞系bRNA-seq数据中找出其中发生融合的序列。由于在细胞系测序数据中,绝大部分融合序列都是虚假融合。因此,这些从测序数据中找出的融合被全部认为是虚假融合,作为虚假融合的训练及测试样本。研究人员从PCAWG中下载了3540个经由实验验证的已知的融合基因及其位点作为真实融合的训练及测试样本。研究人员比较了HVLD与Transformer、bi-LSTM等多种模型分类融合基因的能力,结果表明,HVLD模型在分类真实和虚假融合基因上具有较高的准确度。
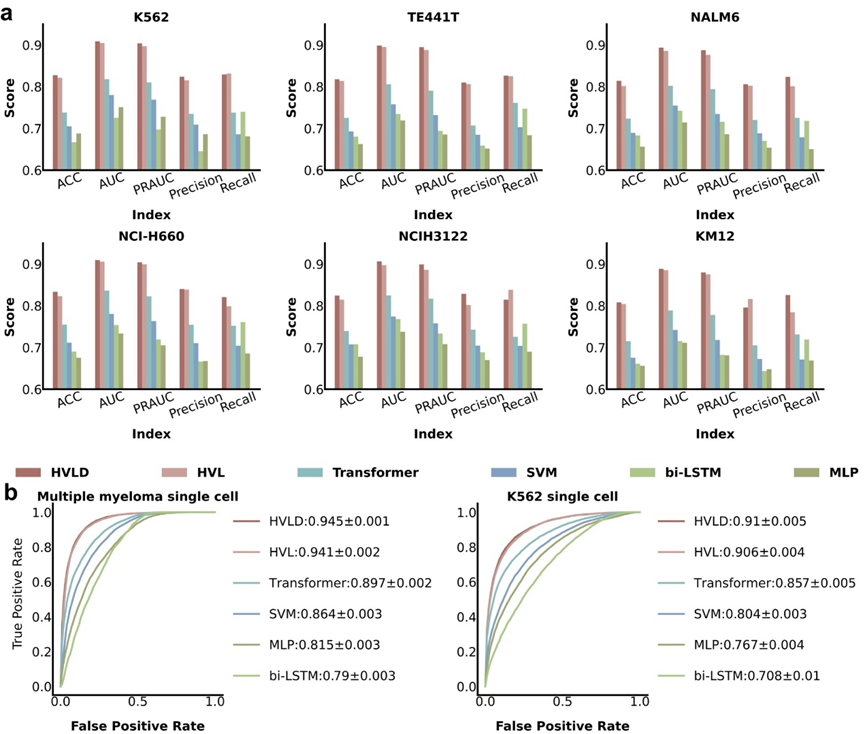
HVDL模型与其他模型在分类真假融合基因上的效果比较
研究人员针对选出的六个融合基因,模拟它们不同情况下的表达水平,并比较Anchored-fusion和其他融合基因检测方法在不同模拟水平下发现跨越读取和分割读取的能力。另外,研究人员也使用真实的肿瘤细胞系bRNA-seq数据,采用不同的下采样比例模拟不同的测序深度,来检验Anchored-fusion在真实细胞系测序数据上的表现。结果表明Anchored-fusion在模拟和真实数据中都能检测到最多的融合基因支持证据。相对其他方法,Anchored-fusion能够在更苛刻的条件下发现融合基因。另外,DUX4基因在染色体上具有数百个拷贝,使得其参与的融合基因成为检测难点。Anchored-fusion在模拟和真实数据中都成功地检测到了CIC-DUX4和DUX4-IGH基因,并且相较其他基因需要更低的测序水平。
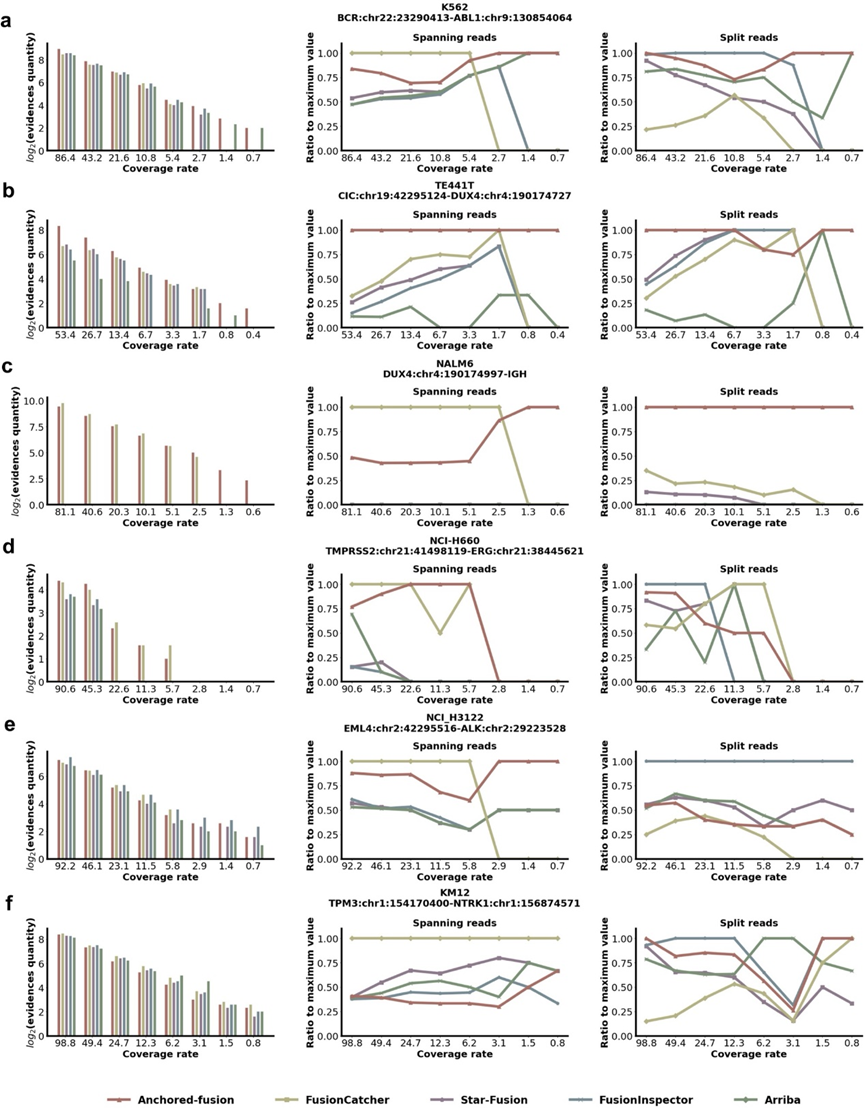
Anchored-fusion及其他方法在bRNA-seq数据中发现融合基因能力的比较
为了探索Anchored-fusion在细胞系单细胞转录组测序数据集中发现融合基因的能力, 研究人员在一个包含350个单细胞的k562细胞系中检测BCR-ABL1融合基因。Anchored-fusion在350个细胞中发现了35个细胞含有BCR-ABL1融合基因,远多于其他方法报告的含有BCR-ABL1融合基因的细胞数目。对于这些单细胞所含有的融合基因支持证据进一步分析表明,Anchored-fusion能够灵敏地检测到具有低水平支持证据的单细胞,而其他方法则很难检测到这类细胞。
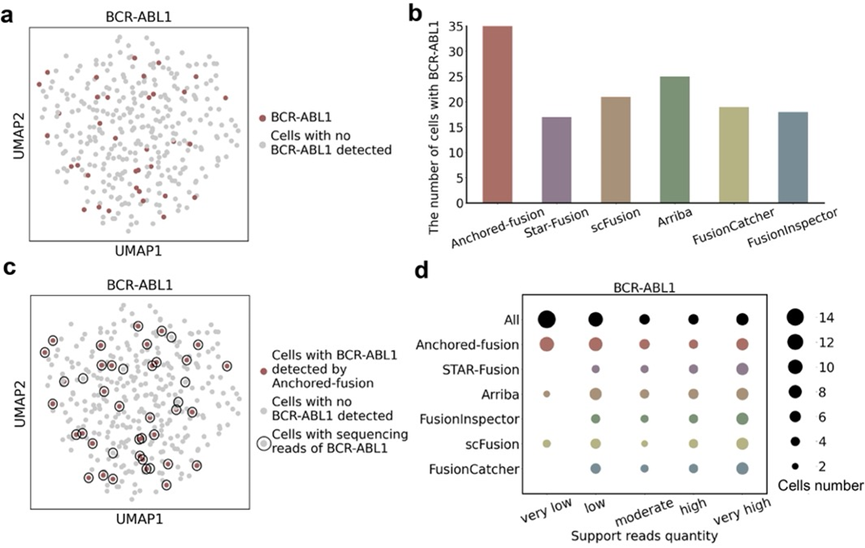
Anchored-fusion 及其他方法在细胞系scRNA-seq数据中发现融合基因能力比较
研究者们评估了Anchored-fusion在临床单细胞RNA-seq数据中发现融合基因的能力。他们选择Jang等人研究的多发性骨髓瘤(MM)单细胞数据集,该数据集包含来自15名患者的597个单细胞,评估Anchored-fusion检测NSD2-IGH融合基因的表现。Anchored-fusion成功发现其中三名患者具有NSD2-IGH融合基因,这与Jang等人的研究结果相同。而其他工具在患者RMM1中发现的具有NSD2-IGH融合基因的细胞数目很少,甚至没有发现。此外,在任何一个病人中,Anchored-fusion都发现了数目最多的目标细胞。这些证据表明,Anchored-fusion的高灵敏性使它比其他方法更适合作为临床水平的单细胞融合基因检测工具。
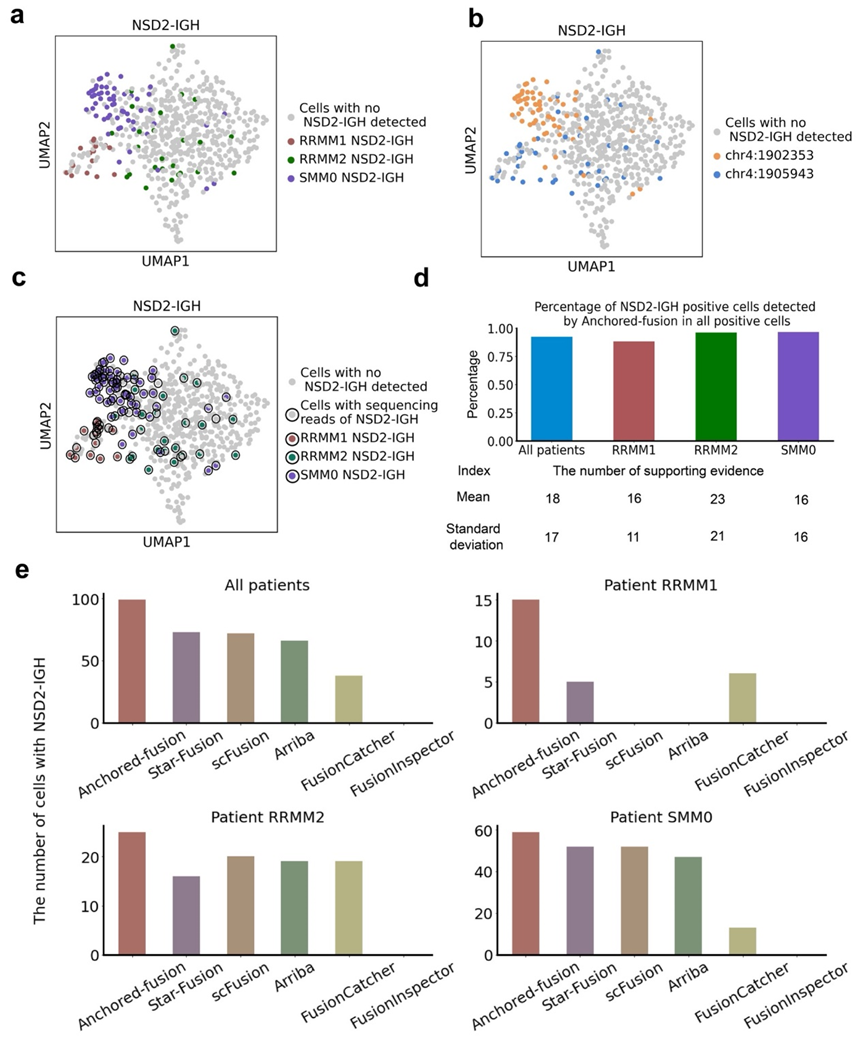
Anchored-fusion及其他方法在临床病人scRNA-seq数据中发现融合基因能力比较
综上,该研究开发了一个高灵敏地检测融合基因的算法工具,并通过各项实验验证了其在模拟测序数据、细胞系bRNA-seq、细胞系scRNA-seq和临床scRNA-seq上的高灵敏性。这些结果都充分证明了Anchored-fusion的高灵敏性,并且具有应用与在临床数据中检测融合基因的可能。
良渚实验室沈宁研究员、浙江大学计算机学院王海帅研究员为本文的通讯作者,联合培养硕士生原溪璐为本文的第一作者。该研究获得了国家重点研发项目 (2022ZD0160703)、国家自然科学基金项目(62202422),浙江省科技计划项目(2022C03134) 、良渚实验室经费的支持。

